Диагностически значимые однонуклеотидные полиморфизмы в локусе env-гена для SNP-генотипирования Bovine leukemia virus

Изучение генетических механизмов формирования патологического процесса ретровирусных инфекций у продуктивных сельскохозяйственных животных и новых подходов к их диагностике и ликвидации — актуальный предмет исследования, выделенный в самостоятельный раздел фундаментальных и поисковых научных исследований (п. 4.3.1.3) по направлению «ветеринария» (п. 4.3.1) на 2021–2030 гг. в Российской Федерации.
Вид Bovine leukemia virus (BLV) семейства Retroviridae — наиболее проблемный биологический объект исследования в ветеринарной лейкозологии, являющийся этиологическим агентом развития персистентного лимфоцитоза и лимфосаркомы у крупного рогатого скота.
Немаловажную роль в патогенезе инфекционного процесса, вызванного BLV, отводят оболочечному (от англ. envelope, сокр. env) env-гену возбудителя, кодирующему в том числе зрелый внеклеточный белок gp51, обладающий как антигенными, так и иммуногенными свойствами с инициацией высокой экспрессии специфических антител у инфицированных животных.
На указанную генетическую мишень и антитела к транслируемому белку нацелены многие ПЦР, ИФА и РИД тест-системы для диагностики лейкоза крупного рогатого скота, а также экспериментальные средства специфической профилактики данного заболевания в виде рекомбинантных вакцин.
Филогенетический анализ секвенируемых нуклеотидных последовательностей локуса env-гена BLV является основным, но не единственным подходом к его генотипической классификации. Базовый способ типизации возбудителя может быть дополнен вспомогательными стратегиями ПЦР-ПДРФ-генотипирования и другими молекулярно-генетическими методами, требующими развития.
Цель исследования — выявление однонуклеотидных полиморфизмов в локусе env-гена Bovine leukemia virus, рассматриваемых в качестве диагностически значимых для SNP-генотипирования BLV на основе анализа данных прямого секвенирования ПЦР-продукта.
Материал и методы исследования
Работа выполнена в лаборатории лейкозологии Всероссийского научно-исследовательского института экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко Российской академии наук в 2023 г.
Теоретико-аналитическая часть биоинформационного исследования по картированию однонуклеотидных полиморфизмов и выявлению диагностически значимых SNPs проведена выравниванием в онлайн-программах BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi) и CLUSTALW (https://www.genome.jp/tools-bin/clustalw) депонированных в GenBank NCBI нуклеотидных последовательностей локуса env-гена 101 типового изолята известных генотипов BLV (https://www.ncbi.nlm.nih.gov/datasets/taxonomy/11901/), ограниченных праймерами env5099 и env5521.
Для практической реализации экспериментальной части исследования по SNP-генотипированию BLV, запланированного в следующей работе, потребуется: постановка с экстрагированными образцами провирусной ДНК «гнездовой» ПЦР с «внешними» (env5032 и env5608) и «внутренними» (env5099 и env5521) праймерами; детекция специфичного ПЦР-продукта длиной 444 bp методом горизонтального электрофореза в 2,5% агарозном геле с последующей визуализацией полученных электрофореграмм в УФ-трансиллюминаторе; секвенирование ампликонов анализируемого локуса env-гена BLV на генетическом анализаторе «НАНОФОР-05» (Синтол, Россия) — восьмиканальном секвенаторе (секвенирование по Сэнгеру) с использованием «внутренних» праймеров в качестве сиквенсных с последующей интерпретацией детектируемых однонуклеотидных полиморфизмов в идентификационном ключе.
Результаты и обсуждение
В результате выравнивания нуклеотидных последовательностей локуса env-гена 101 типового изолята известных генотипов Bovine leukemia virus в общей сложности было выявлено 155 однонуклеотидных полиморфизмов, из которых всего 35 SNPs рассматривались в качестве диагностически значимых (табл. 1).
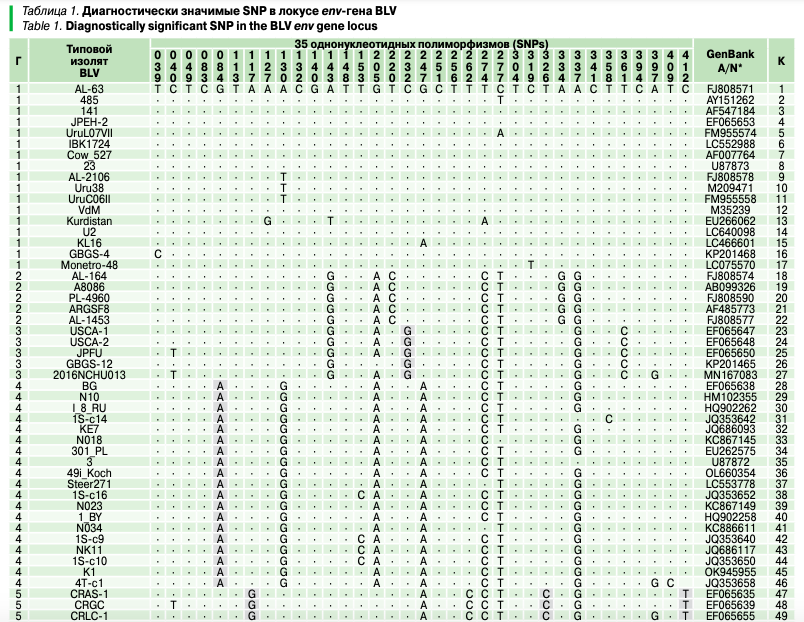
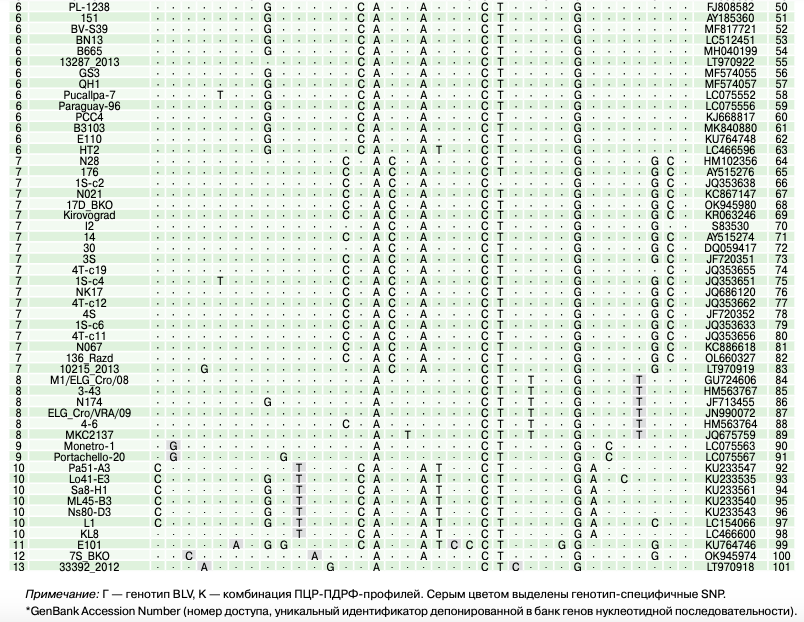
Исходя из данных таблиц 1, 1-й генотип BLV может быть идентифицирован интерпретацией результатов детекции сразу двух SNP (G205G и A337A). При этом анализ по одной только SNP в положении 205 не позволит дискриминировать 1-й генотип от 5-го, а также дифференцировать его представителей от изолята GBGS-12 (GenBank A/N: KP201465) 3-го генотипа BLV, тогда как SNP-анализ по другой только полиморфной позиции 337 не обеспечит полноценной идентификации 1-го генотипа от 4-го из-за двух недифференцируемых изолятов у последнего — 1S-c14 (JQ353642) и 3 (U87872).
Для идентификации 2-го генотипа BLV необходимо учитывать результаты детекции не менее двух SNP (T220С и A334G). При анализе же только одной SNP в положении 220 невозможно будет дифференцировать 2-й генотип от 7-го, а SNP-анализ только полиморфной позиции 334 не позволит дискриминировать 2-й генотип от 11-го генотипа BLV. 3-й и 4-й генотипы BLV имеют по одной генотип-специфичной SNP (C232G и G84A соответственно), благодаря чему могут быть успешно идентифицированы даже при нацеливании лишь на указанные полиморфные позиции. 5-й генотип BLV обладает сразу тремя генотип-специфичными SNPs (A117G, T326C, C412T), что существенно повышает идентификационную способность SNP-генотипирования в отношении данного генотипа при секвенировании амплифицируемого локуса env-гена.
Идентификация 6-го генотипа BLV хоть и затруднена отсутствием у него генотип-специфичной SNP, но выполнима при фокусировании на четыре полиморфные позиции (A127R, C132C, T153C и C341С). По ним невозможно дифференцировать его только от 4-го и 11-го генотипов. Однако благодаря наличию у 4-го и 11-го генотипов BLV генотип-специфичных SNP, проблема их дифференциации фактически решаема. 7-й генотип BLV также не имеет генотип-специфичной SNP, но всё равно может быть идентифицирован при SNP-анализе не менее двух полиморфных позиций (T220C и A334A), в совокупности обеспечивающих его дифференциацию от всех остальных генотипов BLV. 8-й, 9-й и 10-й генотипы BLV характеризуются наличием у них по одной генотип-специфичной SNP (C394T, C40G и C132T соответственно), тем самым гарантируя эффективную процедуру их идентификации при SNP-анализе указанных полиморфных позиций.
Оставшиеся три генотипа BLV имеют по две генотип-специфичные SNP: T113A и T256С у 11-го, T49С и G140A у 12-го, C83A и T304С у 13-го генотипа BLV соответственно. Следовательно, указанные полиморфные позиции являются диагностически значимыми при идентификации перечисленных генотипов BLV. 13-й генотип, занимающий промежуточное положение между 2-м и 3-м, имеет схожую c ними полиморфную позицию (A143G), позволяющую дифференцировать их от других генотипов BLV.
Таким образом для SNP-генотипирования BLV может быть достаточен анализ даже по 22 охарактеризованным однонуклеотидным полиморфизмам, дополнительно представленным в сводной таблице 2.

При этом девять генотипов BLV (3-й, 4-й, 5-й, 8-й, 9-й, 10-й, 11-й, 12-й и 13-й) характеризуются генотип-специфичными SNP и благодаря им эффективно идентифицируются, тогда как четыре генотипа BLV (1-й, 2-й, 6-й и 7-й) не имеют генотип-специфичных SNP, но всё же могут быть идентифицированы при интерпретации результатов детекции целого ряда однонуклеотидных полиморфизмов, диагностически значимых для определенного генотипа.
Выводы
В проанализированной выборке выравненных нуклеотидных последовательностей локуса env-гена типовых изолятов известных генотипов BLV выявлено 155 однонуклеотидных полиморфизмов, из которых 35 SNP рассматривались в качестве диагностически значимых. При этом для SNP-генотипирования BLV на основе анализа данных прямого секвенирования ПЦР-продукта сделан акцент на 22 охарактеризованных однонуклеотидных полиморфизмах, включая 14 генотип-специфичных SNP, обеспечивающих идентификацию девяти генотипов BLV: 3-го (C232G), 4-го (G84A), 5-го (A117G, T326C, C412T), 8-го (C394T), 9-го (C40G), 10-го (C132T), 11-го (T113A и T256С), 12-го (T49С и G140A) и 13-го (C83A и T304С). Другие восемь полиморфных позиций хоть и не являлись генотип-специфичными, но интерпретация результатов их анализа имеет идентификационную ценность в отношении оставшихся четырех генотипов BLV: 1-го (G205G и A337A), 2-го (T220С и A334G), 6-го (A127R, C132C, T153C и C341С) и 7-го ((T220C и A334A).
Об авторах
Рамиль Ришадович Вафин, доктор биологических наук, профессор,Федеральный научный центр — Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко РАН, Рязанский пр-т, 24, Москва, 109428, Россия
vafin-ramil@mail.ru; https://orcid.org/0000-0003-0914-0053
Хамид Халимович Гильманов, кандидат биологических наук, Федеральный научный центр — Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко РАН, Рязанский пр-т, 24, Москва, 109428, Россия
Gilmanov.xx@mai.ru; https://orcid.org/0000-0001-7053-6925
Павел Николаевич Шастин, кандидат ветеринарных наук, Федеральный научный центр — Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко РАН, Рязанский пр-т, 24, Москва, 109428, Россия
shastin.pasha@yandex.ru; https://orcid.org/0000-0001-7360-927X
Евгений Алексеевич Гулюкин, аспирант, Федеральный научный центр — Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко РАН, Рязанский пр-т, 24, Москва, 109428, Россия
gulyukin_viev@mail.ru; https://orcid.org/0000-0001-9898
Артем Геннадьевич Григорьев, младший научный сотрудник, Федеральный научный центр — Всероссийский научно-исследовательский институт экспериментальной ветеринарии им. К.И. Скрябина и Я.Р. Коваленко РАН, Рязанский пр-т, 24, Москва, 109428, Россия
griartis.viev@yandex.ru8 https://orcid.org/0000-0002-9731-5417
УДК 619:57.065:578.828 DOI: 10.32634/0869-8155-2023-372-7-22-26
Сельское хозяйство, ветеринария, зоотехния, агрономия, агроинженерия, пищевые технологии





















